| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- 몬테카를로 학습
- 확률공리
- genetic algorithm
- Traveling salesman problem
- 도커 컨테이너
- 스네이크케이스
- 유전알고리즘
- mmoe
- 산업공학
- 코딩스타일
- 케밥케이스
- Metaheuristic
- Docker Image
- 파스칼케이스
- q learning
- Federated learning
- 강화학습
- 그리드월드
- multi task learning
- 연합학습
- 딥러닝
- on-policy
- 카멜케이스
- 큐러닝
- off-policy
- 도커 개념
- 배반사건
- routing problem
- Sarsa
- 헝가리안노테이션
- Today
- Total
SU Library
[Meta-heuristic] Genetic Algorithm, 유전 알고리즘 본문
정말 오랜만에 글을 쓰네요. 6월 7일 디펜스이후로 열심히 블로그를 하려고했지만, 디펜스이후 치워야될 일들이 너무 불어나는 바람에... 바빠서 현실과 싸우는 중입니다. 그와중에 몇가지 정리된 사안들이 있어서 약간의 여유가 생긴지라, 그동안 배웠던 지식을 복습하고, 다시 정리하는 차원에서 대표적인 메타 휴리스틱 알고리즘인 Genetic Algorithm(GA)에 대해 작성하게 되었습니다. ㅎㅎ 최적화학문에 대한 첫 포스팅인 만큼 메타휴리스틱이 무엇인지, 이걸로 무엇을 할건지에 대해 간략히 설명하고 넘어가겠습니다.
메타 휴리스틱이란?
풀고자하는 문제의 최적해(정답)를 제한된 시간과 한정된 자원으로 풀기위한 알고리즘 입니다. 이는 선형계획모델 등 전통적인 최적화 기법으로 reasonable한 시간안에 풀기 어려운 문제(NP-hard)에 대한 근사해를 도출 할 수 있는 알고리즘입니다. 그중 GA는 매우 유명한 기법중 하나입니다.
Genetic Algorithm의 개념

GA는 말그대로 생물이 자손을 이어감에 따라 아래 그림과 같이 부모가 가진 일부 유전자들이 자식들에게 일부 유전되는 것을 본따서 만든 알고리즘입니다.

이 현상을 공학적인 방법으로 적용시킨 방법론입니다. 몇가지 정의들이 등장합니다.
- chromsome(염색체) : 이는 GA를 통해 새롭게 도출된 각각의 솔루션입니다.
- offspring or children (자손) : 각 세대마다 생기는 솔루션들을 의미합니다.
- fitness (적합도) : 각 염섹체(chromsome)들이 문제를 해결함에 있어서 얼마나 적합한 지를 나타냅니다.
- mutation (돌연변이) : 부모 솔루션을 통해 도출된 자손 솔루션에 변형을 가함으로 부모 솔루션이 가진 특징을 파괴하고 새로운 특성을 갖게합니다. 이는 실제 생물학적인 돌연변이현상이 나타나는 것과 비슷하게 구현됩니다. 이렇게 돌연변이를 생성하는 이유는 local optimal에 머물게 하는것을 탈피하기 위함입니다.
- crossover (교차) : 부모 솔루션이 가진 특징들을 공유하는 과정을 나타냅니다.

Genetic Algorithm의 순서도
- 초기 솔루션(랜덤, 혹은 그리디 방식 등으로 생성됨)을 부모 솔루션으로 설정합니다.
- 초기 솔루션들에 대한 fitness를 계산합니다.
- 초기 솔루션들을 이용하여, 자손 솔루션(새로운 솔루션)들을 생성합니다.
- 생성된 새로운 솔루션들에 대한 적합도를 계산합니다.
- 종료조건확인(Iteration 횟수나 감소 폭 혹은 자손의 적합도나 성능이 개선되지 않는 최대 카운트 등으로 설정할 수 있음)을 확인후 종료 조건 불만족 시 자손 솔루션을 부모솔루션으로 두고 3으로 다시 이동합니다.
- 알고리즘 종료
Genetic Algorithm의 적용 예시
본 포스팅에서 GA를 이용하여 NP-hard 문제인 TSP(traveling salesman problem)을 풀이해나가는 과정을 기술하겠습니다.
우선 관련된 패키지들을 import하고 시각화와 랜덤한 좌표들 생성 및 거리를 나타내는 행렬을 생성하는 함수들을 선언합니다.
import numpy as np
import random
import matplotlib.pyplot as plt
color_list = []
for ci in list(range(0,5)):
no_of_colors=5
color="#"+''.join([random.choice('0123456789ABCDEF') for i in range(6)])
color_list.append(color)
def plot_nodes(city_coord_list,colors=color_list,depot_idx = 0):
fig, ax = plt.subplots(figsize=(20,12))
for x,y in city_coord_list:
ax.plot(x,y,color=colors[0],marker='o', alpha = 0.5)
for i in range(len(city_coord_list)-1):
ax.plot([city_coord_list[i][0],city_coord_list[i+1][0]],[city_coord_list[i][1],city_coord_list[i+1][1]],color="blue")
ax.plot([city_coord_list[0][0],city_coord_list[i+1][0]],[city_coord_list[0][1],city_coord_list[i+1][1]],color="blue")
ax.plot(city_coord_list[depot_idx][0],city_coord_list[depot_idx][1],color='red',marker='o')
def l2_distance(coord1,coord2):
return np.abs(coord1[0]-coord2[0])**2 + np.abs(coord1[1]-coord2[1])**2
def get_adjacency_matrix(coord_city_list):
distance_matrix = np.zeros((len(coord_city_list),len(coord_city_list)))
for n_idx, node_1 in enumerate(coord_city_list):
for n_jdx, node_2 in enumerate(coord_city_list):
distance_matrix[n_idx,n_jdx] = l2_distance(node_1,node_2)
return distance_matrix,list(range(len(coord_city_list)))
def generate_random_coordinates(number_of_nodes):
coord_city_list = []
for n_idx in list(range(number_of_nodes)):
random_x_point = np.random.random()*10 +np.random.random()*10
random_y_point = np.random.random()*10 +np.random.random()*10
coord_city_list.append([random_x_point,random_y_point])
admatrix,node_list= get_adjacency_matrix(coord_city_list)
return admatrix,node_list,coord_city_list이 포스팅에서는 100개의 노드를 생성하고 1번노드부터 100번 노드까지 그냥 순차적으로 연결한 것을 초기솔루션으로 가정하겠습니다.
admatrix,node_list,coord_list = generate_random_coordinates(100)
plot_nodes(coord_list)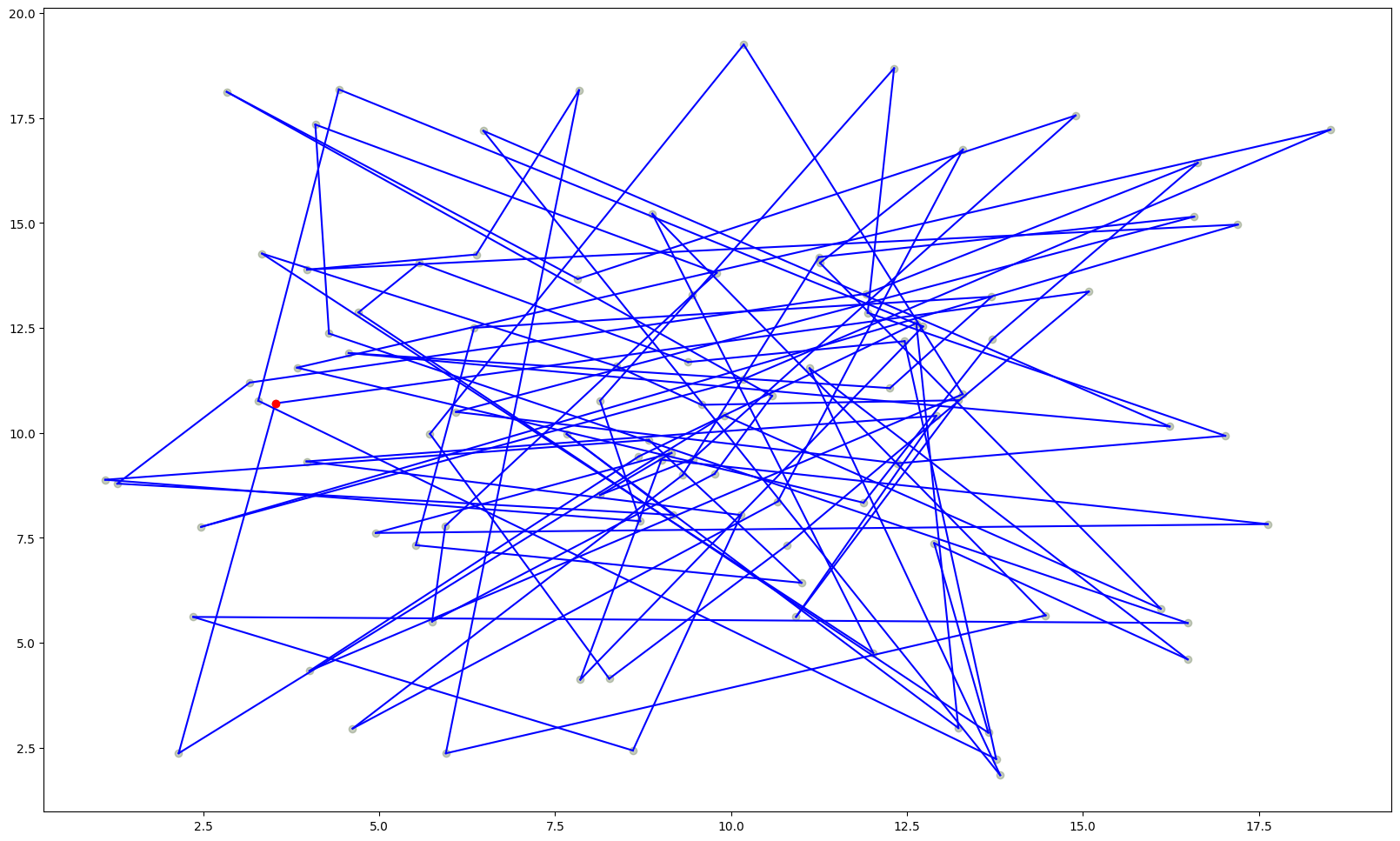
이제 매번 유전자의 정보를 담고, 매 iteration마다 솔루션을 업데이트하기위한 함수를 포함하는 클래스Population을 정의하겠습니다.
클래스 선언시 매개변수인 bag은 부모 솔루션을 생성하는데 쓰이는 유전자 소스들입니다. 클래스 차원에서 거리행렬을 저장하는 이유는 생성된 유전자의 적합성을 평가하기 위함입니다.
class Population():
def __init__(self,bag,admatrix):
self.bag = bag
self.parents = []
self.score = 0
self.best =None
self.adjacency_mat = admatrix
def fitness(self,chromosome):
return sum([
self.adjacency_mat[chromosome[i],chromosome[i+1]]
for i in range(len(chromosome)-1)
])
def evaluate(self):
distances = np.asarray(
[self.fitness(chromosome) for chromosome in self.bag]
)
self.score = np.min(distances)
self.best = self.bag[distances.tolist().index(self.score)]
self.parents.append(self.best)
return distances / np.sum(distances)
def select(self,k=4):
fit = self.evaluate()
while len(self.parents)< k:
idx = np.random.randint(0,len(fit))
if fit[idx] < np.random.rand():
self.parents.append(self.bag[idx])
self.parents = np.asarray(self.parents)
def swap(self,chromosome):
a, b = np.random.choice(len(chromosome), 2)
chromosome[a], chromosome[b] = (
chromosome[b],
chromosome[a],
)
return chromosome
def crossover(self,p_cross = 0.1):
children = []
count,size = self.parents.shape
for _ in range(len(self.bag)):
if np.random.rand() > p_cross:
children.append(
list(self.parents[np.random.randint(count,size=1)[0]])
)
else:
parent1,parent2 = self.parents[np.random.choice(count,size=2),:]
idx = np.random.choice(range(10), size=2, replace=False)
start, end = min(idx), max(idx)
child = [None] * size
for i in range(start, end + 1, 1):
child[i] = parent1[i]
pointer = 0
for i in range(size):
if child[i] is None:
while parent2[pointer] in child:
pointer+=1
child[i] = parent2[pointer]
children.append(child)
return children
def mutate(self, p_cross=0.1, p_mut=0.1):
next_bag = []
children = self.crossover(p_cross)
for child in children:
if np.random.rand() < p_mut:
next_bag.append(self.swap(child))
else:
next_bag.append(child)
return next_bag- fitness : 매개변수로 받은 유전자의 적합성을 평가하기위한 함수입니다. 여기서는 매개변수로 받은 유전자 솔루션의 총 거리를 계산합니다.
- evaluate : 클래스 내에 존재하는 모든 유전자들의 우위를 결정하는 함수입니다.
- select : evaluate함수를 통해 부모 유전자의 개수를 생성합니다. 매개변수로 받은 k의 개수만큼 생성합니다.
- swap : mutate에 사용되는 함수인데, 기존에 생성된 솔루션을 일부 파괴하고 새로 재창조함으로 기존의 부모 유전자가 가진 특성을 지워버리는 함수입니다.
- crossover : 두 부모유전자의 솔루션을 쎃어서 새로운 자손솔루션을 도출하는과정입니다. 여기서각 부모 유전자가 얼마나 어느비율로 쎃일지는 랜덤한 값에 의존합니다. 그 기준을 p_cross라는 매개변수를 통해 어느정도 조절할 수 있습니다.
- mutate : crossover를 통해 생성된 자손 유전자의 값을 확률에따라 swap으로 변형시키는 과정입니다.
아래는 초기 솔루션을 생성하는 함수입니다.
def init_population(cities,adjacency_matrix,n_population):
return Population(
np.asanyarray([np.random.permutation(cities) for _ in list(range(n_population))]),adjacency_matrix
)그리고 이때까지 정의한 코드들을 활용하여 GA의 순서도에 작성한 매커니즘을 담은 함수, 즉 문제를 직접적으로 풀기위한 함수를 정의하겠습니다.
매개변수로 도시 인덱스 -cities,
거리행렬 -adjacency_mat,
매번 얼마나 염색체들을 생성할 것인지 -n_population,
얼마나 GA를 돌릴 것인지 -n_iter,
n_population개수 대비 부모유전자를 얼마나 생성할 것인지 - selectivity 등을 매개변수로 받습니다.
def genetic_algorithm(
cities,
adjacency_mat,
n_population=5,
n_iter=100,
selectivity=0.15,
p_cross=0.5,
p_mut=0.1,
print_interval=100):
pop = init_population(cities,adjacency_mat,n_population=n_population)
best= pop.best
score = float('inf')
history = []
for i in range(n_iter):
pop.select(n_population*selectivity)
history.append(pop.score)
if i%print_interval==0:
print(f"Generation {i}: {pop.score}")
if pop.score <score:
best = pop.best
score = pop.score
children = pop.mutate(p_cross,p_mut)
pop = Population(children,pop.adjacency_mat)
return best,historyinit_population함수를 이용하여 초기 솔루션을 생성합니다. 생성된 초기솔루션 객체값을 담고있는 pop의 best값을 저장하고 이를 베이스로 n_iter횟수만큼 휴리스틱과정을 돌립니다. 크게 다음의 과정을 반복합니다.
- 염색체들을 이용하여 부모 솔루션을 생성합니다.(current solution)
- 생성된 score보다 더 좋은 값을 가졌는지 확인하고 그렇다면 업데이트 합니다.
- 부모 솔루션의 염색체들을 이용하여 children을 생성합니다.
- 생성된 children의 염색체들을 저장하고 이를 활용해서 부모 솔루션을 생성할 준비를 합니다.
아래는 100개의 문제를 풀기위해 설정한 함수를 실행시킵니다.
best,history= genetic_algorithm(node_list, admatrix,n_population=200,
n_iter=2000,print_interval=100)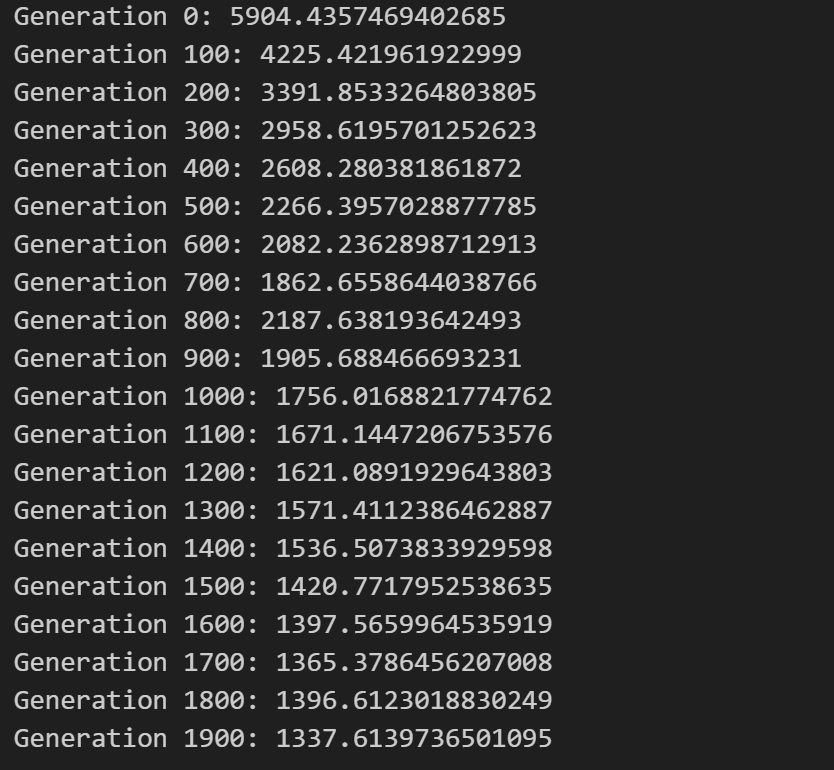
이 history를 간단히 출력해본다면, 다음과 같습니다.
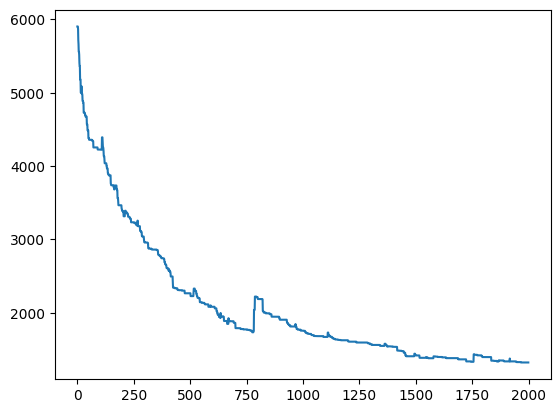
마지막으로 best solution에 대한 결과를 출력한다면!
plot_nodes(np.asarray(coord_list)[best],depot_idx=best.index(0))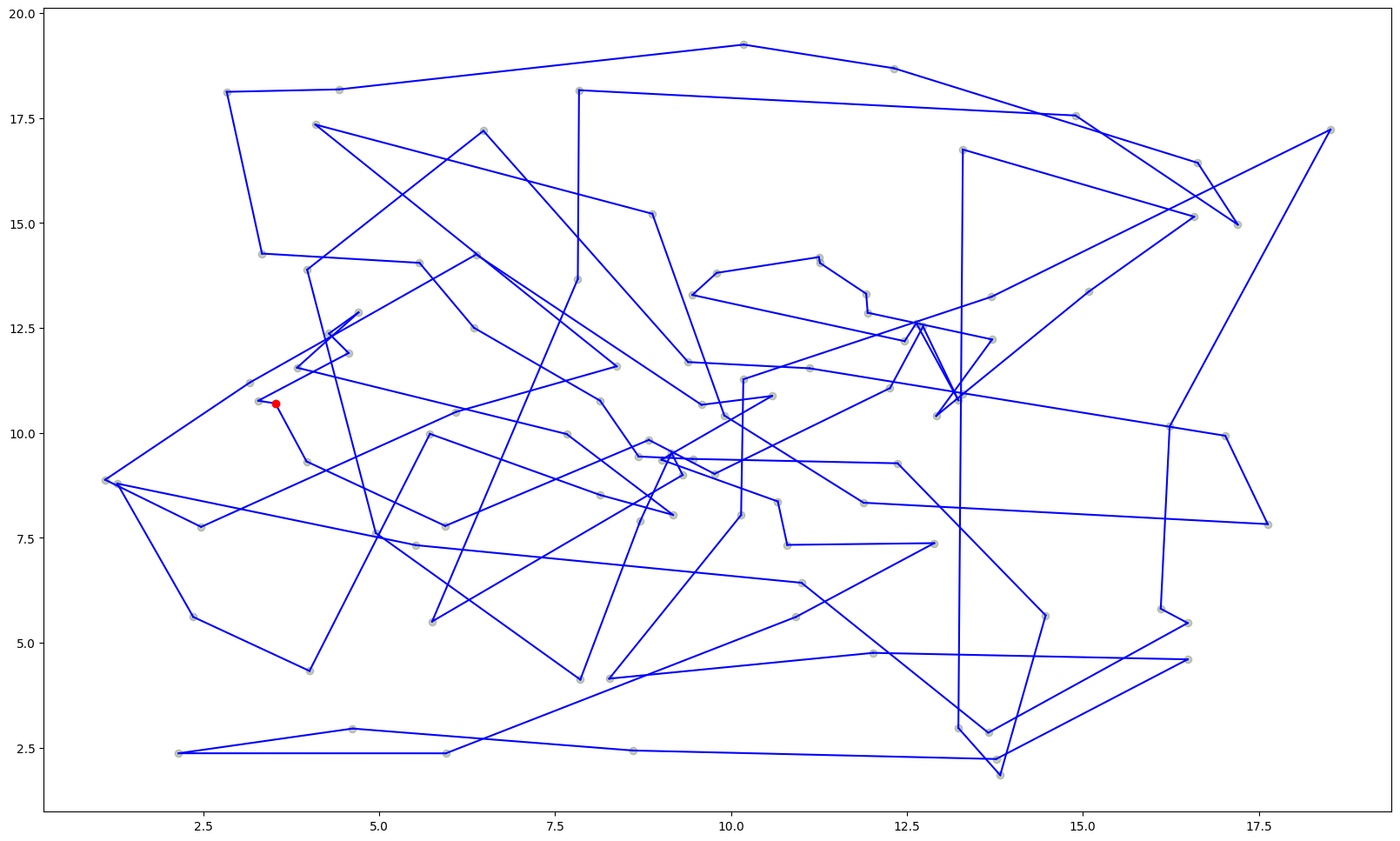
노드의 개수를 20개로 줄였을 시의 결과도 함께 첨부하겠습니다.
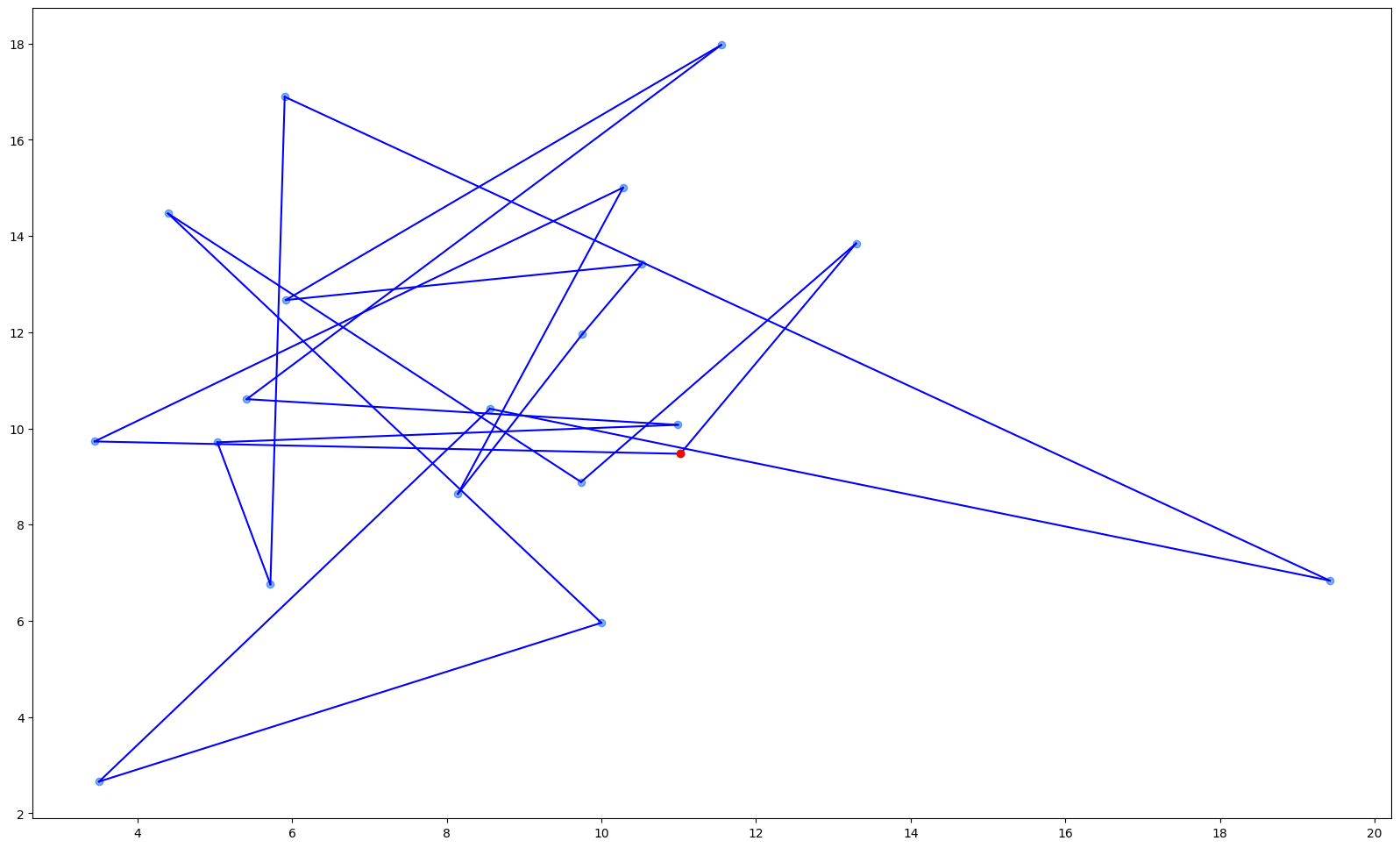
GA를 돌린 결과입니다.
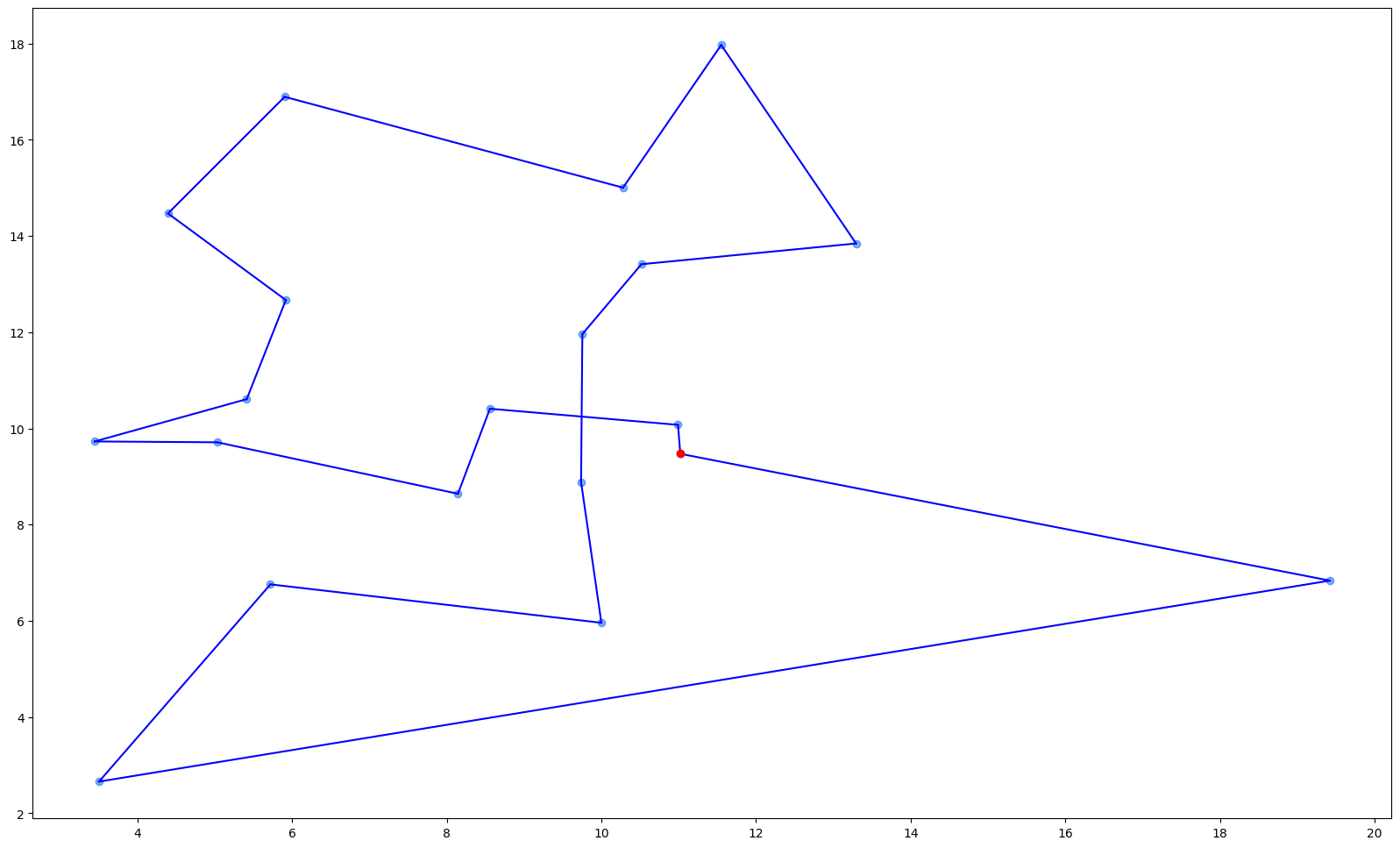
기존 초기 솔루션 대비 상당히 개선된 결과를 확인할 수 있습니다.
이렇게 NP-hard문제를 푸는 GA에 대해 포스팅하는 시간을 가졌습니다. 다음은 선형계획 모델에 대해 어떻게 우리가 환경에대한 수학적인 정의를 하고 최적해를 도출할 수있는지 프로그래밍적인 방법으로 다른 문제 location-allocation문제를 가지고와서 풀어보도록 하겠습니다.
